引言
DFA分析方法是由C.-K提出的一种研究时间序列波动长时相关性的方法。主要用来区别复杂系统本身产生的波动和由外界及环境刺激作用在系统上产生的波动。外部刺激产生的变化假设引起了局部效应,而系统本身内部的动力学产生的变化假设展示了长时的相关性。DFA是尺度不变行为的另一测度方法,因为它估计了所有尺度的显示分形特性的趋势。DFA计算中包括强调由非平稳性引起的相关性减少的局部趋势,和量化代表系统本身性质的长时分形关联性特征。
DFA提供给临床医生一种研究系统本身内在机制产生的生理信号长时相关性的方法,该研究不包括由外界产生的与系统本身不相关的刺激信号。该方法利用整个序列来进行计算而且尺度自由,所以能够提供区别生理信号的有用信息。理论上,尺度指数
从0.5(随机序列)变化到1.5(随机步速),生理信号的尺度指数接近1。大于1的尺度指数表示长时尺度相关性的丢失和机体本身的病理改变。该技术最初应用来探测DNA序列的长时相关性,后来广泛的应用在生理时间序列的分析。
DFA方法已经被利用来估计癫痫的发生与诊断,老年人,癫痫病人都显示了分形尺度的丢失。在心脏研究中通过对2小时ECG纪录进行分析发现,DFA能够提供传统的时域、频域分析所不能提供的信息。在与其他分析方法相比较分析24小时癫痫病发后病人的癫痫数据中,尺度指数的下降,明显的预测了脑部活动失常的猝发。在分析睡眠无呼吸病人的脑部活动变化中DFA方法明显优于谱分析方法。在癫痫病发后预测死亡率的研究中,短时尺度指数的下降是个很好的预测参数。总之,脑部波动信号中DFA尺度指数的变化能够对癫痫的发生和诊断提供辅助诊断和预测信息。
我在本文中运用DFA方法分析了不同生理病理状态的脑电图的自相似特性,得出了一些比较结论,可以对临床诊断有重要的提示作用。
1 原理
脑电图(electroencephalogram,简称EEG)作为脑部生理电活动的图形记录,反映脑部兴奋的产生、传导和恢复过程中的电变化,疾病对脑部神经细胞电兴奋传导的影响也会在脑电图中表现出来,如图一。因此采用脑电图对脑部生理电活动进行检测和分析一直是医学临床实践中心脏功能检测和诊断的最重要方法和手段。

图一 正常与癫痫病人的脑电图
为了克服生理数据时间序列的高度非平稳性,提出了处理随机游走的一种改进的均方根分析方法(去趋势波动分析,简称DFA)以分析生物医学数据的自相似特性。它比传统方法(如频谱分析和Hurst分析)的优点是,它可以检测出一个似乎是非平稳时间序列的内在的自相似性,同时可避免杂散地检测可明显看出来的可能是一个人为的外在趋势自相似性。DFA算法更适用于某些慢变趋势非平稳时间序列。
具体算法如下:
针对如图1所示的某个心电时间序列(总长度为N),首先进行求和

其中,Bi是第i个数据,Bave而是所分析的心电时间序列的平均值。这种求和步骤可以映射原始时间序列到一个自相似的过程。
接着,量度求和后的时间序列的垂直特征尺度。方法是:求和后的时间序列被分成等长为正在上传…重新上传取消的许多个小片段。画出每一个长度为的小片段的最小二乘拟和直线(它用来代表该片段中的趋势)(图2)。直线段的y坐标标记为ynk。

图二 局域的去趋势图示
(垂直线显示片段长度为10000,图中直线段代表着该求和后的时间序列的趋势由对每一个片段进行线性最小二乘拟合画出)
其次,对求和后的时间序列进行去趋势,即在每一个片段中把yk减去局域趋势ynk。对一个给定的片段长度,这种求和并且去趋势后的时间序列的涨落的特征尺寸可以由下式计算:

在所有时间尺度(片段大小)上重复上面的计算,可以得出和片段大小的关系曲线。对的斜率决定尺度指数(自相似参量)。
2 方法
应用DFA方法来分析两种典型的生理时间序列。我们采集了5个健康人、5个癫痫病人的EEG数据.在每组数据里取10000个点,然后计算该时间序列并进行分析。
3 结果
为了进一步探讨DFA与尺度的关系,在这次实验中,我选择了4、8、20、50、100、200、500、1000八个不同的尺度;并且,为了让实验具有普遍性,避免实验初患者的不适应带来的生理反应的影响,我们随机选择了正常人和癫痫患者EEG信号的中部信号。将这部分信号加载进python进行实验。为了研究正常人和癫痫病人的EEG信号,我们不妨选择了C3导联所采集的信号作为主要的研究对象。
实验中DFA分尺度拟合的情况如图三。


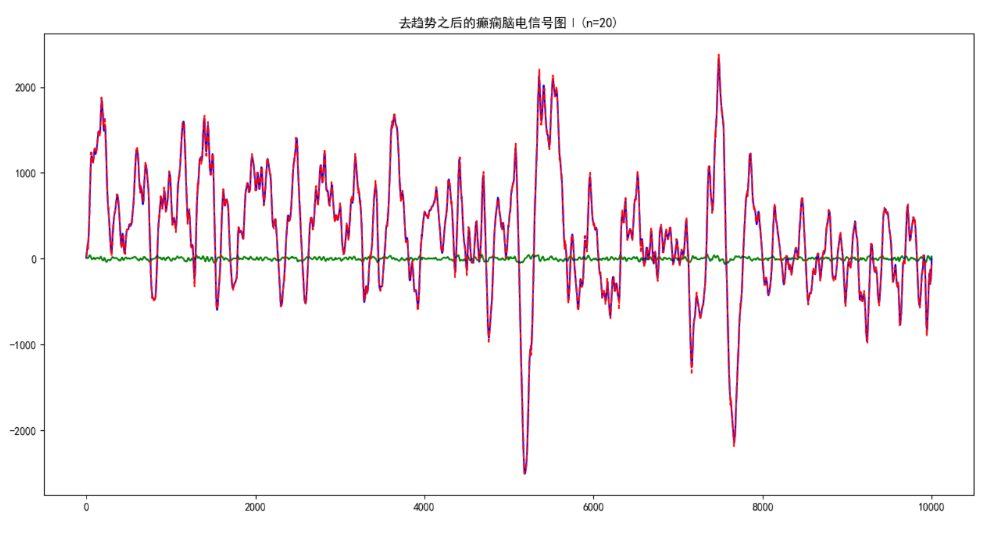


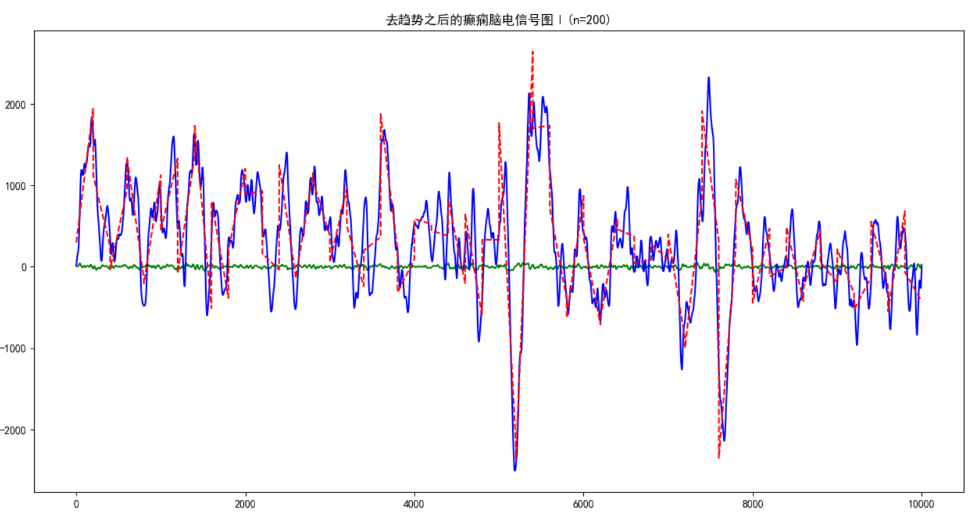


图三 DFA在八种不同尺度下面的分形图和原始EEG图对比
通过DFA的数据分析,得到了斜率决定尺度正在上传…重新上传取消的值,用最小二乘法拟合后得到图四的你和图像。

图四 斜率决定尺度图
通过DFA算法,为了在算法层面上区分正常人和脑电病人EEG信号的区别,我们从散点图上可以看出,癫痫病人的EEG的FN值要比正常人的FN值大且分布散乱,如图五。
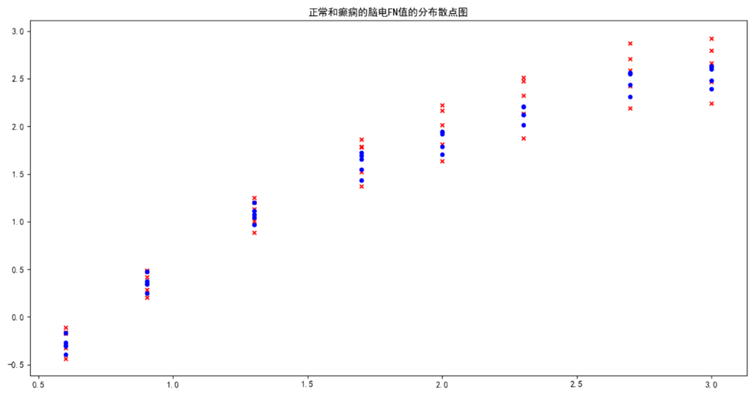
图五 正常人和癫痫病人FN值的分布散点图
在图六中可以清楚的看到,癫痫病人的DFA指数,即斜率决定尺度(自相似参量),最大值高于正常人,最小值低于正常人,并且自相似参量的分布非常的散乱,数值跨度大。因此,DFA算法可以明显的区分癫痫患者和正常患者,在临床诊断上能给医学工作者带来一定的辅助用途,降低误诊率。
4 讨论与结论
我们用DFA的方法研究了不同生理病理状态下的心电图的自相似特性变化情况,结果说明对于指征癫痫疾病演化的趋势,DFA是一个比较好的方法。
研究发现,从医学统计来说,心电图的正在上传…重新上传取消均值随着脑部神经健康状况的变差呈先下降后上升趋势。心电图的DFA值波动范围,健康人的比较稳定在某个范围之内;而冠心病人的值变动范围比较大;心梗病人的变动范围最大。研究表明的均值和波动范围的变动情况可以比较有效地揭示脑部健康状况,的均值和波动范围的变动情况可以比较有效地揭示心脏健康状况,尤其是值波动范围的大小变化是一个早期发现癫痫疾病较为灵敏的参数,具有临床诊断意义。
5 代码
读取文件的函数open_file.py
import random
import codecs
def open_file(file_path):
f = codecs.open(file_path)
line = f.readline()
data = []
data1 = []
real_data1 = []
while line:
a = line.split()
b = a[4:5]
b1 = ','.join(map(str, b))
data.append(b)
data1.append(b1)
line = f.readline()
data.remove(['C3'])
data1.remove('C3')
real_data = [float(a) for a in data1]
r = round(random.uniform(0, 1), 2) * 5000
for i in range(int(r), int(r) + 10000):
real_data1.append(real_data[i])
return real_data1
数据预处理分割字符串的函数split_list.py
def list_of_groups(init_list, children_list_len):
list_of_groups = zip(*(iter(init_list),) *children_list_len)
end_list = [list(i) for i in list_of_groups]
count = len(init_list) % children_list_len
end_list.append(init_list[-count:]) if count !=0 else end_list
return end_list
数据处理函数data_process.py
import split_list
import math
import numpy as np
def process(variable_name,group_count):
ave_value = []
variable_sum = 0
for i in range(0, len(variable_name)):
variable_sum += variable_name[i]
ave = variable_sum / len(variable_name)
for i in range(0, len(variable_name)):
ave_value.append(variable_name[i] - ave)
yi = np.cumsum(ave_value) # yi是文献中的y(k)
temp = []
for i in range(0, len(variable_name)):
temp.append(i)
x_data1 = split_list.list_of_groups(temp, group_count)
y_data1 = split_list.list_of_groups(yi, group_count) # y_data1 = yi
temp1 = []
for i in range(0, int(len(variable_name)/group_count)):
poly = np.polyfit(x_data1[i], y_data1[i], deg=1)
temp2 = np.polyval(poly, x_data1[i])
for j in range(0, group_count):
temp1.append(temp2[j])
y_data2 = split_list.list_of_groups(temp1, group_count) # 去趋势之后的函数值 yk
yk_minus = []
t = []
a = 0
for i in range(0, int(len(variable_name)/group_count)):
yk_minus.append(list(map(lambda x: x[0] - x[1], zip(y_data1[i], y_data2[i]))))
for j in range(0, group_count):
t.append(yk_minus[i][j])
for i in range(0,len(t)):
a += (t[i])**2
fn = math.sqrt(a / len(t))
return temp,temp1,yi,fn
画图 DFA_Signal_figure.py
from open_file import open_file
from matplotlib import pyplot as plt
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
from data_process import *
import math
fn = []
epilepsy_1 = open_file('E:/DFA/DFA_DATA/癫痫异常脑电图/20121030105258.txt')
epilepsy_2 = open_file('E:/DFA/DFA_DATA/癫痫异常脑电图/20121031103719.txt')
epilepsy_3 = open_file('E:/DFA/DFA_DATA/癫痫异常脑电图/20121031111404.txt')
epilepsy_4 = open_file('E:/DFA/DFA_DATA/癫痫异常脑电图/20121031113934.txt')
epilepsy_5 = open_file('E:/DFA/DFA_DATA/癫痫异常脑电图/20121031163308.txt')
normal_1 = open_file('E:/DFA/DFA_DATA/正常脑电图/20121030084615.txt')
normal_2 = open_file('E:/DFA/DFA_DATA/正常脑电图/20121030091121.txt')
normal_3 = open_file('E:/DFA/DFA_DATA/正常脑电图/20121030103952.txt')
normal_4 = open_file('E:/DFA/DFA_DATA/正常脑电图/20121030110908.txt')
normal_5 = open_file('E:/DFA/DFA_DATA/正常脑电图/20121030151450.txt')
plt.plot(epilepsy_1, label='abnormal_1 plot')
plt.plot(epilepsy_2, label='abnormal_2 plot')
plt.plot(normal_1, label='normal plot')
# generate a legend box
plt.legend(bbox_to_anchor=(0., 1.02, 1., .102), loc=0,ncol=3, mode="expand", borderaxespad=0.)
plt.show()
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,4)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=4)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,8)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=8)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,20)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=20)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,50)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=50)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,100)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=100)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,200)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=200)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,500)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=500)')
plt.show()
fn.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(epilepsy_1,1000)
plt.plot(temp_1,epilepsy_1,'g',temp_1,yk_1,'b',temp_1,fitted_value_1,'r--')
plt.title('去趋势之后的癫痫脑电信号图Ⅰ(n=1000)')
plt.show()
fn.append(fn_1)
number = [4,8,20,50,100,200,500,1000]
number_log = []
fn_log = []
for i in range(0,len(number)):
number_log.append(math.log(number[i],10))
fn_log.append(math.log(fn[i],10))
poly = np.polyfit(number_log, fn_log, deg = 1)
plt.plot(number_log, fn_log,'o')
plt.plot(number_log, np.polyval(poly,number_log))
plt.show()
fn_normal_1 = []
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,4)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,8)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,20)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,50)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,100)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,200)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,500)
fn_normal_1.append(fn_1)
temp_1,fitted_value_1,yk_1,fn_1 = process(normal_1,1000)
fn_normal_1.append(fn_1)
#
# fn_normal_log = []
# for i in range(0,len(number)):
# fn_normal_log.append(math.log(fn_normal_1[i],10))
# poly = np.polyfit(number_log, fn_log, deg = 1)
# plt.plot(number_log, fn_normal_log,'o')
# plt.plot(number_log, np.polyval(poly,number_log))
#
DFA_scatter_figure.py
from matplotlib import pyplot as plt
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
from DFA_signal_figure import *
import math
FN_abnormal = []
FN_normal = []
member_count = [4,8,20,50,100,200,500,1000]
group_name_abnormal = [epilepsy_1,epilepsy_2,epilepsy_3,epilepsy_4,epilepsy_5]
group_name_normal = [normal_1,normal_2,normal_3,normal_4,normal_5]
for i in range(0,len(group_name_abnormal)):
for j in range(0,len(member_count)):
temp_1, fitted_value_1, yk_1, fn_1 = process(group_name_abnormal[i], member_count[j])
FN_abnormal.append(fn_1)
for i in range(0,len(group_name_normal)):
for j in range(0,len(member_count)):
temp_1, fitted_value_1, yk_1, fn_1 = process(group_name_normal[i], member_count[j])
FN_normal.append(fn_1)
dimension_4_abnormal = []
dimension_8_abnormal = []
dimension_20_abnormal = []
dimension_50_abnormal = []
dimension_100_abnormal = []
dimension_200_abnormal = []
dimension_500_abnormal = []
dimension_1000_abnormal = []
dimension_4_normal = []
dimension_8_normal = []
dimension_20_normal = []
dimension_50_normal = []
dimension_100_normal = []
dimension_200_normal = []
dimension_500_normal = []
dimension_1000_normal = []
x = [0,8,16,24,32]
for i in range(0,len(x)):
dimension_4_abnormal.append(math.log(FN_abnormal[x[i]],10))
dimension_8_abnormal.append(math.log(FN_abnormal[x[i] + 1],10))
dimension_20_abnormal.append(math.log(FN_abnormal[x[i] + 2],10))
dimension_50_abnormal.append(math.log(FN_abnormal[x[i] + 3],10))
dimension_100_abnormal.append(math.log(FN_abnormal[x[i] + 4],10))
dimension_200_abnormal.append(math.log(FN_abnormal[x[i] + 5],10))
dimension_500_abnormal.append(math.log(FN_abnormal[x[i] + 6],10))
dimension_1000_abnormal.append(math.log(FN_abnormal[x[i] + 7],10))
dimension_4_normal.append(math.log(FN_normal[x[i]],10))
dimension_8_normal.append(math.log(FN_normal[x[i] + 1 ],10))
dimension_20_normal.append(math.log(FN_normal[x[i] + 2 ],10))
dimension_50_normal.append(math.log(FN_normal[x[i] + 3 ],10))
dimension_100_normal.append(math.log(FN_normal[x[i] + 4 ],10))
dimension_200_normal.append(math.log(FN_normal[x[i] + 5 ],10))
dimension_500_normal.append(math.log(FN_normal[x[i] + 6 ],10))
dimension_1000_normal.append(math.log(FN_normal[x[i] + 7 ],10))
log_x = []
for i in range(len(member_count)):
log_x.append(math.log(member_count[i],10))
x1 = []
x2 = []
x3 = []
x4 = []
x5 = []
x6 = []
x7 = []
x8 = []
for i in range(0,5):
x1.append(math.log(4, 10))
x2.append(math.log(8,10))
x3.append(math.log(20, 10))
x4.append(math.log(50, 10))
x5.append(math.log(100, 10))
x6.append(math.log(200, 10))
x7.append(math.log(500, 10))
x8.append(math.log(1000, 10))
plt.scatter(x1, dimension_4_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x2, dimension_8_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x3, dimension_20_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x4, dimension_50_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x5, dimension_100_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x6, dimension_200_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x7, dimension_500_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x8, dimension_1000_abnormal, marker = 'x',color = 'red', s = 20 ,label = 'First')
plt.scatter(x1, dimension_4_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x2, dimension_8_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x3, dimension_20_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x4, dimension_50_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x5, dimension_100_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x6, dimension_200_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x7, dimension_500_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.scatter(x8, dimension_1000_normal, marker = 'o',color = 'blue', s = 20 ,label = 'First')
plt.title('正常和癫痫的脑电FN值的分布散点图')
plt.show()
# plt.plot(epilepsy_1, label='abnormal_1 plot')
# plt.plot(epilepsy_2, label='abnormal_2 plot')
# plt.plot(normal_1, label='normal plot')
# # generate a legend box
# plt.legend(bbox_to_anchor=(0., 1.02, 1., .102), loc=0,ncol=3, mode="expand", borderaxespad=0.)
# # annotate an important value
# plt.annotate("Important value", (55, 20), xycoords='data',xytext=(5, 38),arrowprops=dict(arrowstyle='->'))
# plt.show()
boyplot.py
import numpy as np
from DFA_scatter_figure import FN_normal,FN_abnormal
import pandas as pd
import matplotlib.pyplot as plt
import math
logFN_normal = []
logFN_abnormal = []
for i in range(0,len(FN_normal)):
logFN_normal.append(math.log(FN_normal[i],10))
for i in range(0,len(FN_abnormal)):
logFN_abnormal.append(math.log(FN_abnormal[i],10))
normal = []
abnormal = []
for i in range(0,len(FN_abnormal)):
if i%8 == 0 :
normal.append(float(logFN_normal[i])/math.log(4,10))
abnormal.append(float(logFN_abnormal[i])/math.log(4,10))
if i%8 == 1:
normal.append(float(logFN_normal[i]) / math.log(8, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(8, 10))
if i%8 == 2:
normal.append(float(logFN_normal[i]) / math.log(20, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(20, 10))
if i%8 == 3:
normal.append(float(logFN_normal[i]) / math.log(50, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(50, 10))
if i%8 == 4:
normal.append(float(logFN_normal[i]) / math.log(100, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(100, 10))
if i%8 == 5:
normal.append(float(logFN_normal[i]) / math.log(200, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(200, 10))
if i%8 == 6:
normal.append(float(logFN_normal[i]) / math.log(500, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(500, 10))
if i%8 == 7:
normal.append(float(logFN_normal[i]) / math.log(1000, 10))
abnormal.append(float(logFN_abnormal[i]) / math.log(1000, 10))
print(normal)
print(abnormal)
data = {
'normal': normal,
'abnormal': abnormal,
}
df = pd.DataFrame(data)
df.plot.box(title="正常与非正常情况下DFA指数的分布图")
plt.grid(linestyle="--", alpha=0.3)
plt.show()